tcj-cimat2024
Taller de Ciencia para Jóvenes 2024 CIMAT. Descubriendo la diversidad microbiana a través del ADN
Sesión 2 Algoritmos en biología (50 min)
Un algoritmo es un proceso con pasos finito que tiene un principio y un final. Por ejemplo cambiar una llanta es un algoritmo. En Biología se aplican mucho algoritmos matemáticos y computacionales. La Evolución Biológica es el conjunto de cambios en poblaciones a través de generaciones. Este proceso ha generado la diversidad de especies en la Tierra. Varios algoritmos deben aplicarse para lograr visualizar los procesos evolutivos, incluyendo el cálculo de distancia entre secuencias de ADN.
🔎 Explora la aplicación de Evolución del Museo Virtual de Matemáticas.
Distancia entre dos secuencias
La distancia mas simple entre dos secuencias es un 0 si tienen la misma base o un 1 si no la tienen. Mira el siguiente ejemplo
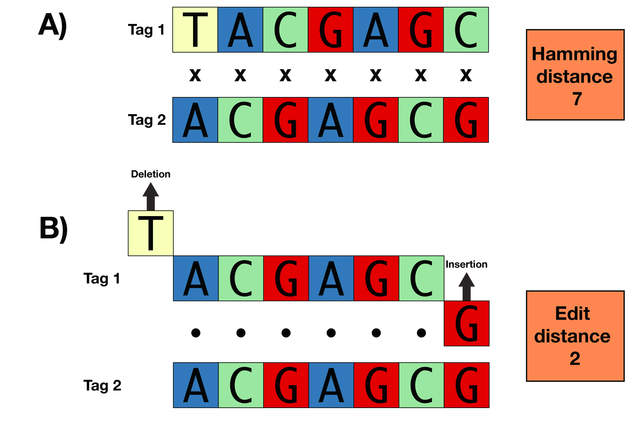
¿Qué pasa aquí?
Cuál es la distancia de Hamming entre la secuencia 1 y las demás secuencias??
seq1
CAGGACCACACA
seq2
CAGGACCACACA
seq3
CACGACCACACA
seq4
CGGACCACACA
¿Qué distancia te gustaría que fuera?
Algoritmos de alineamiento
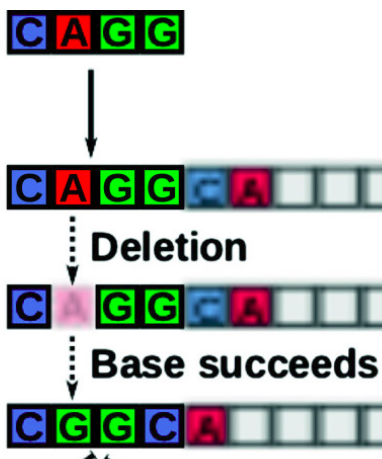
Como podemos ver, eventos evolutivos como las deleciones e inserciones crean la necesidad de alinear secuencias para poder obtener un mejor estimado de sus distancias evlutivas.
Ejemplo 1
seq1 CAGGACCACACA
seq3 CA-GACCACACA
Ejemplo 2
El 16s es una de las secuencias más conservadas, vamos a hacer un alineamiento entre las secuencias de 16s de estos organismos. Vamos a alinearlos:
>NR_112305.1_Streptomyces_coelicolor_12854_16S_ribosomal_RNA ACGAACGCTGGCGGCGTGCTTAACACATGCAAGTCGAACGATGAACCGCTTTCGGGCGGGGATTAGTGGC GAACGGGTGAGTAACACGTGGGCAATCTGCCCTGCACTCTGGGACAAGCCCTGGAAACGGGGTCTAATAC CGGATATGACTGTCCATCGCATGGTGGATGGTGTAAAGCTCCGGCGGTGCAGGATGAGCCCGCGGCCTAT CAGCTTGTTGGTGAGGTAGTGGCTCACCAAGGCGACGACGGGTAGCCGGCCTGAGAGGGCGACCGGCCAC ACTGGGACTGAGACACGGCCCAGACTCCTACGGGAGGCAGCAGTGGGGAATATTGCACAATGGGCGAAAG CCTGATGCAGCGACGCCGCGTGAGGGATGACGGCCTTCGGGTTGTAAACCTCTTTCAGCAGGGAAGAAGC GAAAGTGACGGTACCTGCAGAAGAAGCGCCGGCTAACTACGTGCCAGCAGCCGCGGTAATACGTAGGGCG CAAGCGTTGTCCGGAATTATTGGGCGTAAAGAGCTCGTAGGCGGCTTGTCACGTCGGTTGTGAAAGCCCG GGGCTTAACCCCGGGTCTGCAGTCGATACGGGCAGGCTAGAGTTCGGTAGGGGAGATCGGAATTCCTGGT GTAGCGGTGAAATGCGCAGATATCAGGAGGAACACCGGTGGCGAAGGCGGATCTCTGGGCCGATACTGAC GCTGAGGAGCGAAAGCGTGGGGAGCGAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGGTGGGC ACTAGGTGTGGGCAACATTCCACGTTGTCCGTGCCGCAGCTAACGCATTAAGTGCCCCGCCTGGGGAGTA CGGCCGCAAGGCTAAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGCGGAGCATGTGGCTTAATTC GACGCAACGCGAAGAACCTTACCAAGGCTTGACATACACCGGAAACGTCTGGAGACAGGCGCCCCCTTGT GGTCGGTGTACAGGTGGTGCATGGCTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACG AGCGCAACCCTTGTCCCGTGTTGCCAGCAGGCCCTTGTGGTGCTGGGGACTCACGGGAGACCGCCGGGGT CAACTCGGAGGAAGGTGGGGACGACGTCAAGTCATCATGCCCCTTATGTCTTGGGCTGCACACGTGCTAC AATGGCCGGTACAATGAGCTGCGATACCGCGAGGTGGAGCGAATCTCAAAAAGCCGGTCTCAGTTCGGAT TGGGGTCTGCAACTCGACCCCATGAAGTCGGAGTCGCTAGTAATCGCAGATCAGCATTGCTGCGGTGAAT ACGTTCCCGGGCCTTGTACACACCGCCCGTCACGTCACGAAAGTCGGTAACACCCGAAGCCGGTGGCCCA ACCCCTTGTGGGAGGGAGCTGTCGAAGGTGGGACTGGCGATTGGGACGAAGTCGTAACAAGGTAGCCGTA CCGGAA
>LC026160.1_Streptomyces_lividans_AS2_gene_for_16S_ribosomal_RNA GGGGAGCTCTGCAGTCGACGATGACCACTTCGGTGGGGATTAGTGGCGAACGGGTGAGTAACACGTGGGC AATCTGCCCTGCACTCTGGGACAAGCCCTGGAAACGGGGTCTAATACCGGATACTGACCCTCGCAGGCAT CTGCGAGGTTCGAAAGCTCCGGCGGTGCAGGATGAGCCCGCGGCCTATCAGCTTGTTGGTGAGGTAATGG CTCACCAAGGCGACGACGGGTAGCCGGCCTGAGAGGGCGACCGGCCACACTGGGACTGAGACACGGCCCA GACTCCTACGGGAGGCAGCAGTGGGGAATATTGCACAATGGGCGAAAGCCTGATGCAGCGACGCCGCGTG AGGGATGACGGCCTTCGGGTTGTAAACCTCTTTCAGCAGGGAAGAAGCGAAAGTGACGGTACCTGCAGAA GAAGCGCCGGCTAACTACGTGCCAGCAGCCGCGGTAATACGTAGGGCGCAAGCGTTGTCCGGAATTATTG GGCGTAAAGAGCTCGTAGGCGGCTTGTCGCGTCGGTTGTGAAAGCCCGGGGCTTAACCCCGGGTCTGCAG TCGATACGGGCAGGCTAGAGTTCGGTAGGGGAGATCGGAATTCCTGGTGTAGCGGTGAAATGCGCAGATA TCAGGAGGAACACCGGTGGCGAAGGCGGATCTCTGGGCCGATACTGACGCTGAGGAGCGAAAGCGTGGGG AGCGAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGGTGGGCACTAGGTGTGGGCAACATTCCA CGTTGTCCGTGCCGCAGCTAACGCATTAAGTGCCCCGCCTGGGGAGTACGGCCGCAAGGCTAAAACTCAA AGGAATTGACGGGGGCCCGCACAAGCGGCGGAGCATGTGGCTTAATTCGACGCAACGCGAAGAACCTTAC CAAGGCTTGACATACACCGGAAAGCATCAGAGATGGTGCCCCCCTTGTGGTCGGTGTACAGGTGGTGCAT GGCTGTCGTCAACTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGTCCCGTGGT GGCCACCAGGCCCTTGGGGTGCTGGGGAACTCACGGGAAAACCGCCGGGGTCAAATCCGAAGGAAAGTGG GGGACGACGTCAAAGTCTTCATTGCCCCTTATGGTCTTGGGCTGGCCACCGTGGCTACAAATGGCCCGGT ACAAATGAACTTGCGATACCCGCCAGGGTGGAAGCGAAACCTCAAAAAGCCCGGTTCCAATTCCNGAATG GGGGGCTTCGAACCTCACCCCCCTGGAAAGTCCGAGATCCCCTAGGAATCCGGAAATACCCATTTTGCGC GGGGTAAAACATTTTCCCGCGGCCTTTTTTACACACGCCGCCCTTAAACTCTCAAGAAATTCTTGTACAC CCCCCAGGAACCGGGTGGCCCCCACCCTCTTTTGGGGGAAGGGACTGTTCCCAAAGGTGGGGTTCCCGGC ACTGGGGGGGGAATNGTTTTCCTCAAGAAGGTAA
Alineador embl/muscle Algunos alineadores computacionales no aceptan espacios en los nombres o identificadores muy largos. Puedes modificar el identificador como necesites para poder alinear.
Haz tu propio algoritmo
Algoritmos en linux, lección de loops
Lectura ¿Qué son los modelos de Markov?
Discute este artículo con tus compañeros
Algoritmos de ensamblado
Cuando se te entrega un genoma secuenciado genoma este viene en muchos pedazos de tamaño relacionado a la tecnología utilizada para secuenciar. Para poder encontrar genes y comparar tu genoma con otros genomas, hace falta ensamblarlo. Existen algoritmos para hacer esto desde la terminal, nosotros haremos un ejercicio práctico.
Ejercicio
🔨 To do (Agregar lecturas) Ensambla tu genoma y encuentra a quién pertenece. ¿Qué profundidad tiene? ¿De qué tamaño son tus pedazos en promedio? ¿Cómo le fue a tu compañero? Discute los resultados y anótalos en el documento colaborativo