clavigenomics
Clavigenomics
Clavigenomics es el repositorio del backend de la aplicación de genómica bacteriana comparativa Clavisual.

Clavigenomics está empacado en un contenedor de docker y a través de Clavisual provee las siguientes funcionalidades:
-Calculo de genes comunes en bacterias relacionadas al tomate.
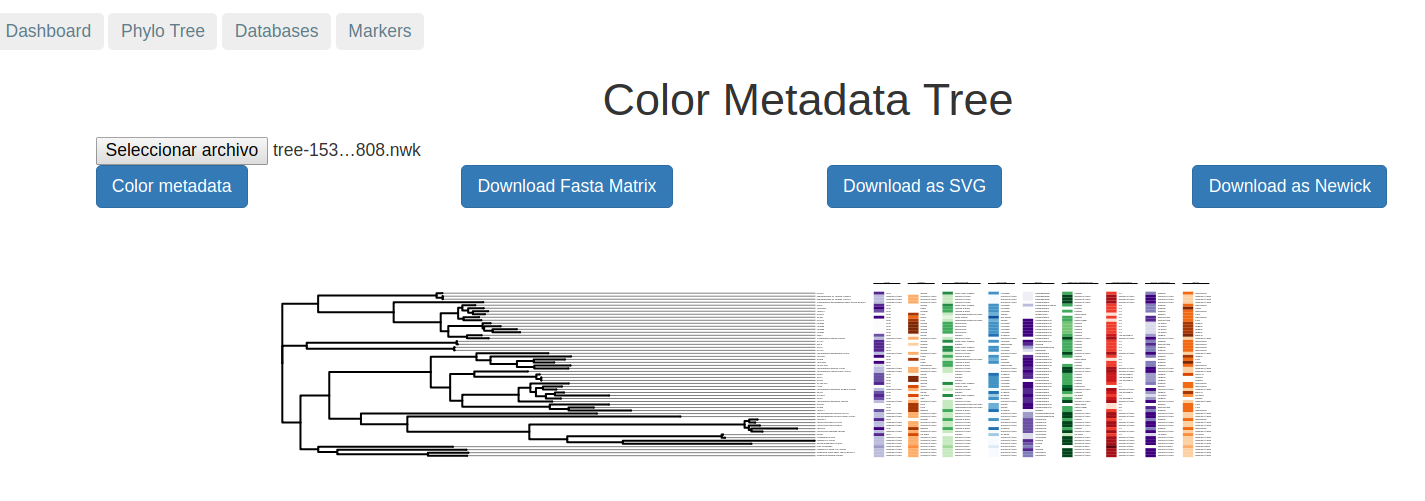
-Visualizacin de árboles filogenéticos con metadatos.
-Cálculo de marcadores de gruops selectos de bacterias relacionadas al tomate.
Aquí se muestra un ejemplo de reconstrucción filogenética visualizada con metadatos y construida con la matriz de genes conservados.
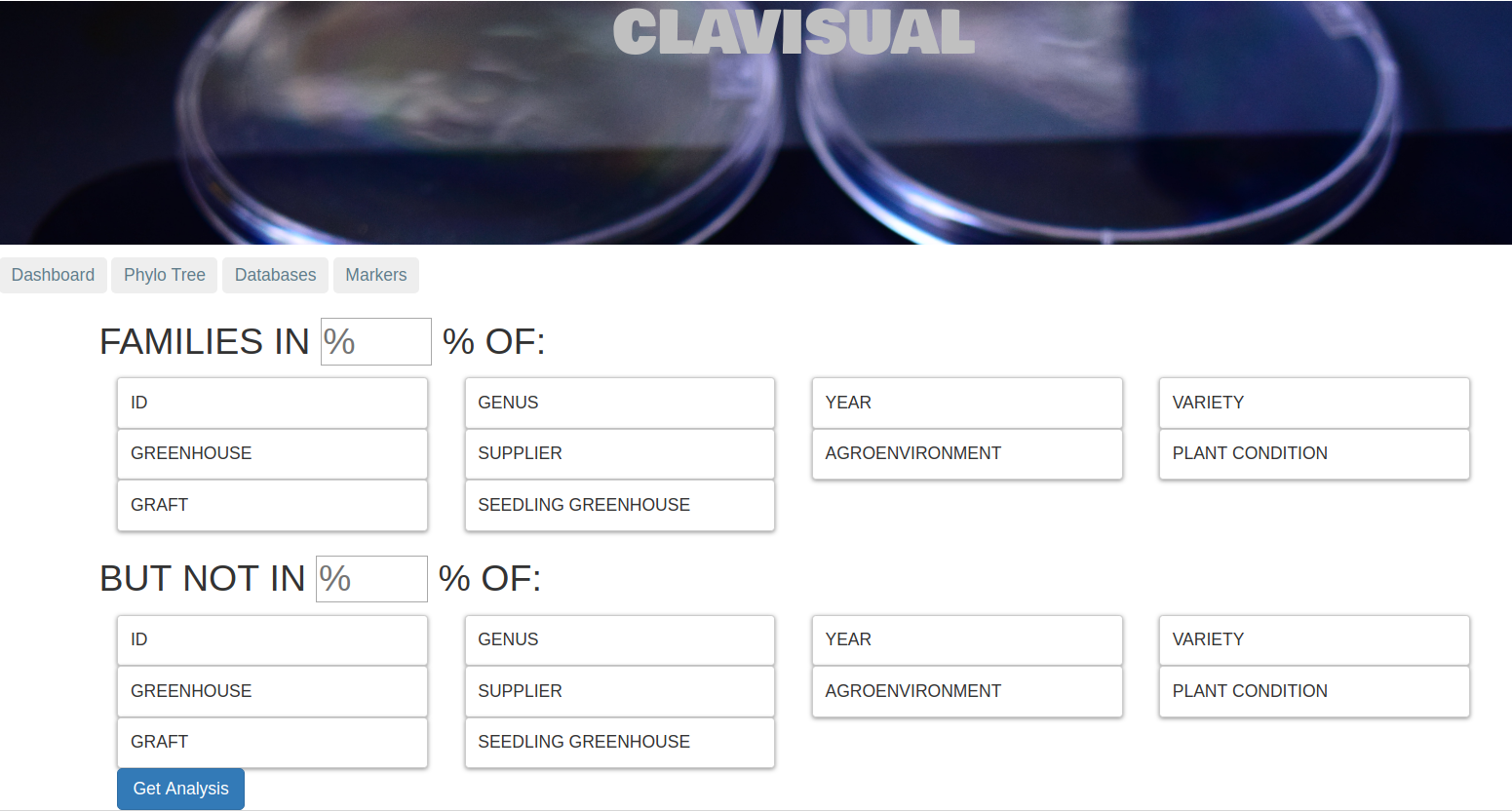
Esta es la interfaz de usuario para el cálculo de grupos de genes marcadores.
Funcionamiento en clavisual
A partir de la sección 3 de este documento todos los archivos de IDs contienen solo el RAST genome id:
6666666.1298689
6666666.1298474
El archivo de Ids sección 1:descargar genomas de RAST y precalcular blast necesita tambien el job id, por requerimiento de myrast.
Ejemplo
876438 6666666.138469
358438 2.1140
Usuarios, por favor, tengan cuidado de escribir los últimos ceros, 2.1140 en lugar de 2.114 como a veces los trata excel.
1 Correr la imagen de docker de modo interactivo
docker run -i -t -v $(pwd):/usr/src/CLAVIGENOMICS nselem/clavigenomic /bin/bash
2. Introducir un nuevo genoma a la aplicación
2.1 Descargar txt faa fna
docker run -i -t -v $(pwd):/home nselem/myrast getFiles.pl <NewIdsFile> <user> <password>
NewIdsFile
876438 6666666.138469
358438 2.1140
Output *txt *faa *fna
2.2. Crear Central (Pseudocore files)
docker run -i -t -v $(pwd):/usr/src/CLAVIGENOMICS nselem/clavigenomic BBH.pl 558ParaCORE <NewIdsFile>
Output *Central
Note: 558ParaCORE is located in /root/clavigenomics/Pseudocore/558ParaCORE
2.3. Crear blast (Contra todos los genomas)
docker run -i -t -v $(pwd):/usr/src/CLAVIGENOMICS nselem/clavigenomic blasting.pl <NewIdsFile> <OldIdsFile>
Output *blast
3. Obtener el Pseudocore
-Correr el PseudoCore
docker run -i -t -v $(pwd):/usr/src/CLAVIGENOMICS nselem/clavigenomic Pseudocore.pl <IdsFile>
Output: Salida.tre FastTree con los ortólogos compartidos concatenados.
realSequences Archivo con el numero de ortólogos realmente compartidos de los 558 propuestos.
Para un ejemplo, mueve los archivos del folder /root/clavigenomics/example/ a /usr/src/CLAVIGENOMICS
cp /root/clavigenomics/example/* .
4. Obtener presencia y ausencia hasta cierto porcentaje de familias
cp /root/clavigenomics/exampleFO/* .
makeFamiliesN.pl /usr/src/CLAVIGENOMICS/ <100> <Presencia> <100> <Ausencia>
Output: temp.n_familias.js
DetailFamilies.pl <FamilyID> /usr/src/CLAVIGENOMICS
ORTHOMCL5018.fna Fasta de nucleotidos
ORTHOMCL5018.tre FastTree de la familia
ORTHOMCL5029.fasta Fasta de proteinas